-
Kill Dogma Revolution !.
www.nature.com/articles/ncomms5945
Pubblicato: 17 settembre 2014
Materiali programmabili basati su biofilm da nanofibre curli ingegnerizzate
Astratto
Il ruolo significativo dei biofilm nella patogenicità ha stimolato la ricerca a prevenire la loro formazione e promuovere la loro distruzione, con conseguente opportunità trascurate di sviluppare biofilm come piattaforma biologica sintetica per materiali funzionali autoassemblanti. Qui presentiamo Biofilm-Integrated Nanofiber Display (BIND) come strategia per la programmazione molecolare del materiale della matrice extracellulare batterica aggiungendo geneticamente domini peptidici alla proteina amiloide CsgA, il componente proteico dominante in Escherichia colibiofilm. Queste proteine di fusione CsgA ingegnerizzate vengono secrete con successo e si autoassemblano a livello extracellulare in reti di nanofibre amiloidi che mantengono le funzioni dei domini peptidici visualizzati. Mostriamo l'uso di BIND per conferire diverse funzioni artificiali alla matrice del biofilm, come la biotemplazione delle nanoparticelle, l'adesione del substrato, l'immobilizzazione covalente delle proteine o una loro combinazione. BIND è una piattaforma nanobiotecnologica versatile per lo sviluppo di materiali robusti con funzioni programmabili, che dimostra il potenziale dell'utilizzo dei biofilm come biomateriali progettabili su larga scala.
introduzione
I progressi nella nostra comprensione dei sistemi batterici nel secolo scorso hanno ampliato il ruolo del microbo dall'essere considerato esclusivamente come una minaccia per la salute all'essere sfruttato come una fabbrica geneticamente programmabile per la produzione di biomolecole e sostanze chimiche. Riteniamo che i biofilm batterici intraprendano una traiettoria simile rispetto ai materiali funzionali avanzati. La maggior parte dei batteri nel mondo naturale esiste come biofilm: comunità organizzate di cellule incastonate in una rete di matrice extracellulare (ECM) composta da polisaccaridi, proteine, acidi nucleici e altri componenti biomolecolari 1. Questo ECM autogenerato protegge i batteri dai rigori ambientali e media l'adesione del substrato, promuovendo così la persistenza microbica e la patogenicità. Pertanto, la maggior parte della ricerca sui biofilm si è concentrata sulla loro eradicazione a causa dei ruoli negativi che i biofilm svolgono nell'infezione clinica 2 .
Prevediamo invece l'addomesticamento dei biofilm come una piattaforma per nanomateriali extracellulari autoassemblanti programmabili e modulari, con il batterio che funge da fonderia vivente per la sintesi di mattoni grezzi, il loro assemblaggio in strutture di ordine superiore dopo la secrezione e il mantenimento del materiale nel suo insieme nel tempo. Sebbene siano state condotte alcune indagini sull'uso dei biofilm per scopi benefici come la generazione di energia 3 , il trattamento delle acque reflue 4 e le biotrasformazioni 5 , 6, questi studi si sono concentrati principalmente sull'alterazione della popolazione dei consorzi microbici di biofilm piuttosto che sul materiale ECM stesso. Un altro esempio è il recente lavoro entusiasmante del gruppo Wood, in cui descrivono la progettazione di circuiti genetici sintetici che modulano l'equilibrio della popolazione in un biofilm a doppia specie per controllare la formazione e la dispersione del biofilm in base al quorum-sensing 7 .
Il nostro approccio alla progettazione del materiale ECM del biofilm per applicazioni pratiche si concentra sul sistema curli, il principale componente strutturale proteico dei biofilm di E. coli . I ricci sono nanofibre amiloidi funzionali altamente robuste con un diametro di circa 4-7 nm che esistono come reti aggrovigliate estese che incapsulano le cellule. I ricci sono formati dall'autoassemblaggio extracellulare di CsgA, una piccola proteina secreta di 13 kDa. Una proteina omologa della membrana esterna, CsgB, nuclea l'assemblaggio di CsgA e ancora le nanofibre alla superficie batterica. Le fibre curli distaccate possono anche esistere come componenti strutturali non associati alle cellule dell'ECM. I geni curli esistono come due operoni trascritti in modo divergente ( csgBAC e csg DEFG ) 8, i cui sette prodotti mediano la struttura (CsgA), la nucleazione (CsgB), l'elaborazione (CsgE, F), la secrezione (CsgC, G) e la regolazione trascrizionale diretta (CsgD) delle nanofibre curli.
Il sistema curli presenta numerose caratteristiche che lo rendono una piattaforma ideale per il tipo di ingegneria dei materiali attraverso la biologia sintetica che immaginiamo. Innanzitutto, poiché la nanofibra curli è composta principalmente dall'autoassemblaggio di una piccola proteina, presenta un punto di ingresso trattabile verso la creazione di una grande diversità di matrici extracellulari di biofilm con metodi di ingegneria genetica convenzionali. Al contrario, sarebbe più difficile progettare il componente esopolisaccaride dei biofilm, poiché la sintesi dei polisaccaridi è spesso legata a percorsi a più fasi con una tolleranza limitata per monomeri chimicamente diversi rispetto al macchinario di sintesi proteica. In secondo luogo, le fibre amiloidi funzionali formate da CsgA sono estremamente robuste, essendo in grado di resistere all'ebollizione nei detergenti 9e l'incubazione estesa in solventi, aumentando la loro potenziale utilità in ambienti difficili. Nanofibre amiloidi simili hanno dimostrato di avere una resistenza paragonabile all'acciaio e una rigidità meccanica paragonabile alla seta 10 , suggerendo che i biofilm con un alto contenuto di amiloide sarebbero in grado di resistere ad ambienti meccanicamente impegnativi. Terzo, le fibrille amiloidi funzionali sono abbondanti in molti biofilm batterici naturali e possono costituire fino al 10-40% del biovolume totale di un biofilm 11, indicando che curli può essere ingegnerizzato artificialmente per comprendere una porzione significativa del biofilm. Inoltre, sebbene analoghi amiloidi funzionali extracellulari siano prodotti da molti batteri, il sistema curli è attualmente il più studiato ed è nativo del batterio modello canonico E. coli , rendendolo un'interessante piattaforma di partenza per lo sviluppo di materiali ingegnerizzati. Infine, recenti scoperte hanno dimostrato che il sistema curli può essere utilizzato per esportare in modo efficiente polipeptidi nativamente spiegati ed era in grado di produrre un frammento di anticorpo camelide funzionale, suggerendo che il sistema curli può essere utilizzato in modo ampio e modulare per la visualizzazione di vari funzionali peptidi in tutto il biofilm di E. coli ECM 12 , 13 .
Qui vi mostriamo un sistema Biofilm-Integrated Nanofiber Display (BIND) per consentire una precisa programmazione genetica del materiale ECM del biofilm di E. coli fondendo domini peptidici funzionali sulla proteina CsgA. Dimostriamo che le varianti chimeriche di CsgA sono secrete dal macchinario di esportazione cellulare nativo e si assemblano in reti di fibre curli che ricordano il sistema wild-type. Mostriamo anche che questa tecnica è compatibile con un'ampia gamma di domini peptidici di varie lunghezze e strutture secondarie. Infine, dimostriamo che i domini peptidici mantengono la loro funzione dopo la secrezione e l'assemblaggio e conferiscono funzioni artificiali al biofilm nel suo insieme. Molto recentemente, Chen et al . 14hanno dimostrato un sistema parallelo basato su curli simile al nostro concetto BIND e mostrano patterning multiscala controllato di singole fibre amiloidi e l'uso di curli ingegnerizzati per l'organizzazione di nanoparticelle d'oro e punti quantici per applicazioni di nanoelettronica. Qui, espandiamo le funzioni che possono essere ingegnerizzate nelle nanofibre curli dimostrando tre ampie funzioni che introduciamo artificialmente nell'ECM del biofilm di E. coli : modellazione di nanoparticelle inorganiche, adesione specifica del substrato abiotico e immobilizzazione covalente sito-specifica di un arbitrario funzionalizzato proteina ricombinante.
Risultati
Determinazione di un sito di fusione peptidico ottimale per CsgA
La strategia BIND consiste nella riprogrammazione del componente amiloide autoassemblante dei biofilm di E. coli utilizzando fusioni genetiche di CsgA a peptidi funzionali ( Fig. 1 ). Per determinare punti di fusione adatti per aggiungere peptidi a CsgA, abbiamo generato una libreria ( Fig. 2a ) costituita da fusioni N- e C-terminali a un dominio peptidico di prova designato MBD (per Metal Binding Domain). È stato dimostrato che il peptide MBD si lega fortemente alle superfici in acciaio inossidabile ed è derivato da un segmento del pilus 15 di Pseudomonas aeruginosa di tipo IV. Le fusioni CsgA-terminale sono state scelte per consentire l'integrazione di peptidi sia lineari che vincolati circolarmente. Sono state preparate tre varianti per ogni capolinea con lunghezze variabili del linker flessibile glicina-serina. Abbiamo usato il colorante colorimetrico di colorazione amiloide standard, Congo Red 16 (CR), per determinare l'entità della produzione di curli per i vari mutanti. Le varianti csgA sono state espresse nel modello E. coli ceppo di delezione csgA LSR10 (MC4100::Δ csgA ) che mantiene il rimanente macchinario per la lavorazione dei curli sotto il regolamento nativo 8 . Questo ceppo è stato precedentemente utilizzato in numerosi studi sul sistema curli in quanto è stato dimostrato che non produce flagelli, cellulosa o lipopolisaccaride O-polisaccaridi, che lo rendono ideale per studi di complementazione dei curli 17 , 18 , 19 . Pertanto, LSR10 è stato scelto come ceppo modello per lo sviluppo di BIND in parte perché qualsiasi segnale colorimetrico ottenuto dalla colorazione CR potrebbe essere attribuito alla presenza di fibre curli prodotte in modo eterologo, al contrario della cellulosa o di altri componenti del biofilm che possono avere la capacità di legare CR in modo non specifico 20 . Allo stesso modo, qualsiasi fibra extracellulare osservata dalla microscopia elettronica a trasmissione (TEM) e dalla microscopia elettronica a scansione (SEM), la caratterizzazione ultrastrutturale può essere attribuita esclusivamente all'autoassemblaggio di mutanti di fusione CsgA ingegnerizzati in modo eterologo.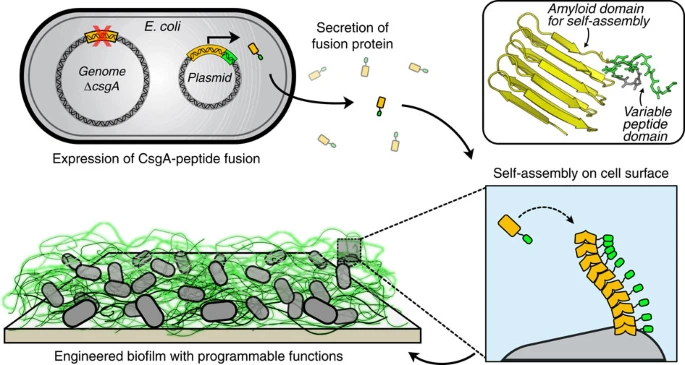
Nella piattaforma BIND, le cellule ΔcsgA esprimono e secernono in modo eterologo proteine di fusione costituite da un dominio amiloidogenico (CsgA, mostrato in arancione) e un dominio funzionale del peptide (verde). Questa proteina di fusione secreta si autoassembla in una rete extracellulare di nanofibre amiloidi ancorate alla superficie cellulare, dando vita a un materiale biofilm con funzioni non naturali programmate. Viene mostrato un modello proteico tridimensionale dei domini peptidici autoassemblanti e funzionali, utilizzando l'infilatura di proteine del modello di omologia della sequenza CsgA su una struttura AgfA. Un esempio di dominio peptidico, SpyTag (vedi Tabella 1 ), è mostrato in verde e il linker flessibile a sei residui in grigio. La struttura del peptide è stata prevista utilizzando PepFold e tutte le manipolazioni strutturali eseguite in PyMol.
Figura 2: Ingegneria genetica della piattaforma BIND.
( a ) Una libreria di mutanti di fusione CsgA in cui l'inserto peptidico MBD (viola) è stato posizionato all'estremità N o C dei domini di ripetizione di curlin (rosso) e affiancato da un linker a sei residui, un linker a due residui o nessun linker (verde). ( b ) La libreria di inserti MBD è stata trasformata in cellule LSR10 (MC4100, ΔcsgA ) e strisciata su piastre a induzione YESCA-Congo Red. La colorazione rossa indica la produzione di amiloide. ( c ) Un insieme rappresentativo di macchie di coltura di una libreria BIND composta da 12 vari peptidi funzionali su piastre di induzione YESCA-Congo Red (enumerate nella Tabella 1 ). ( d) I valori quantitativi di Congo Red sono stati ottenuti da colture quadruplicate YESCA-CR spotted utilizzando misurazioni di quantificazione dell'intensità (ImageJ) dell'amiloide relativa prodotta per ciascuna fusione CsgA-peptide, normalizzata a CsgA wild-type. Viene visualizzato l'errore standard. ( e ) ELISA di filtrazione a cellule intere utilizzando un anticorpo anti-CsgA (barre nere); i controlli di soli anticorpi secondari sono mostrati come barre grigie. Ogni esperimento è stato eseguito in triplicato e viene mostrato l'errore standard. Immagini FE-SEM della libreria BIND di fusione peptidica trasformata in cellule LSR10 (MC4100, Δ csgA ) prive di CsgA ( f ), CsgA wild-type ( g ) e del pannello peptidico BIND (vedi Tabella 1 ): HIS ( h ), GBP ( i ), BANDIERA (j ), CNBP ( k ), A3 ( l ), CLP12 ( m ), QBP1 ( n ), SpyTag ( o ), MBD ( p ), CT43 ( q ), AFP8 ( r ) e Mms6 ( s ). Tutte le barre della scala sono 1 μm.
Il saggio di colorazione CR della libreria di inserimento MBD ha indicato che solo il sito di fusione C3 con il linker C-terminale più lungo tra CsgA e MBD è stato in grado di formare una quantità apprezzabile di fibre amiloidi ( Fig. 2b ), sebbene in una quantità inferiore rispetto al normale -tipo CsgA. È possibile che le fusioni N-terminali abbiano alterato la secrezione a causa della loro vicinanza alla sequenza di riconoscimento dell'esportazione specifica di CsgG. La caratterizzazione ultrastrutturale mediante SEM ( Fig. 2n ) e TEM ( Figure supplementari 1k e 2j ) delle nanofibre curli mutanti C3 ha confermato che mostravano una morfologia simile a quella delle fibre CsgA wild-type.
Le fusioni di peptidi CsgA mantengono la funzione di autoassemblaggio dell'amiloide
Dopo aver identificato il sito di fusione C3 come adatto per la visualizzazione di peptidi sullo scaffold CsgA, abbiamo quindi creato una libreria di 12 diverse fusioni di domini peptidici per testare l'effetto della lunghezza e della struttura del peptide sulla secrezione e sull'assemblaggio. I membri della libreria, dettagliati nella Tabella 1 , variano in lunghezza da 7-59 amminoacidi e codificano un'ampia varietà di funzioni come il legame a vari substrati inorganici (GBP, CNBP, QBP, MBD e AFP8), la nucleazione di nanostrutture minerali e metalliche (A3, CLP12, CT43 e Mms6), e un'interazione catalitica altamente specifica con una proteina (SpyTag) 15 , 21 , 22 , 23 , 24 , 25 , 26 , 27 ,28 , 29 , 30 , 31 . Ciascun dominio peptidico è stato clonato come fusioni C-terminale a CsgA con un linker flessibile di sei aminoacidi interposto ( Tabella 1 ) e questi plasmidi sono stati espressi in cellule LSR10 per produrre 12 diversi biofilm di BIND.
Le differenze relative nella produzione di curli tra i membri della libreria sono state monitorate misurando l'intensità di colorazione dei trasformanti individuati sulle piastre CR ( Fig. 2c,d). Il legame CR è altamente indicativo di amiloidi, fornendo una valutazione qualitativa del materiale extracellulare che lega CR. Questi saggi CR indicano che piccole fusioni peptidiche possono essere tollerate dal macchinario di esportazione curli e potrebbero assemblarsi con successo in reti amiloidi extracellulari come evidenziato da una maggiore colorazione CR di tutte le fusioni peptidiche rispetto al controllo plasmidico vuoto. L'unico mutante per il quale non c'era colorazione CR positiva era il dominio della proteina Mms6 di 59 aminoacidi, confermando i risultati precedenti secondo cui i polipeptidi con sequenze lunghe o struttura intrinseca potrebbero non essere esportati in modo efficiente attraverso il trasportatore della membrana esterna CsgG, che ha una dimensione dei pori di 2 nm (rif. 32). Poiché è noto che CR si lega anche ad altre molecole oltre agli amiloidi, una valutazione più specifica della natura amiloide delle proteine è fornita dalla birifrangenza del materiale colorato con CR osservato sotto luce polarizzata 33 , 34 , 35 . Le masse cellulari delle colonie BIND delle piastre YESCA-CR sono state analizzate mediante microscopia di polarizzazione ( Figura 3 supplementare). La maggior parte delle varianti di BIND mostrava una birifrangenza caratteristica degli amiloidi, sebbene l'intensità della birifrangenza variasse. Il controllo no curli e i campioni Mms6-BIND non hanno mostrato birifrangenza, come previsto. Sorprendentemente, i campioni CLP12- e QBP-BIND hanno mostrato livelli molto bassi di birifrangenza, sebbene abbiano alti livelli di legame CR. Ipotizziamo che ciò sia dovuto all'elevata dispersione delle fibre amiloidi o alla presenza di questi peptidi che alterano la modalità di legame del CR in modo tale da sopprimere la birifrangenza. Per convalidare che la colorazione CR è dovuta alla presenza di materiali extracellulari contenenti CsgA, come le fibre curli, abbiamo eseguito ELISA di filtrazione su cellule intere utilizzando anticorpi anti-CsgA ( Fig. 2e). Solo il materiale extracellulare composto da CsgA e trattenuto dal filtro da 0,22 μm genererebbe un segnale CsgA-positivo. I dati ELISA a cellule intere sono correlati ai risultati della colorazione CR, confermando che le proteine contenenti CsgA sono presenti a livello extracellulare e sono presenti come assemblaggi ad alto peso molecolare. Tuttavia, i dati ELISA non discriminano le proteine di fusione intatte da possibili prodotti di degradazione. Per studiare la possibilità che questi amiloidi extracellulari siano dovuti alla secrezione e all'assemblaggio di proteine di fusione CsgA parzialmente proteolizzate e non delle proteine di fusione CsgA a lunghezza intera desiderate, abbiamo isolato le frazioni extracellulari delle colonie BIND indotte e sottoposto questa frazione a solubilizzazione SDS. La frazione insolubile SDS è stata raccolta mediante ultracentrifugazione e disciolta in esafluoroisopropanolo (HFIP) per disassemblare le fibre curli nei loro componenti monomerici. L'analisi di desorbimento/ionizzazione laser assistita da matrice (MALDI)–time of flight (TOF)/TOF dei campioni disciolti risultanti conferma la presenza di picchi di massa che sono correlati con le proteine di fusione mature CsgA previste in tutti i campioni ad eccezione del no curli controllo e il campione Mms6-BIND (Figure supplementari 4 ). Sebbene ciò non escluda potenziali eventi di proteolisi, dimostra che la frazione extracellulare contiene, in una certa misura, proteine di fusione CsgA intatte che sono in uno stato insolubile SDS, suggerendo che le proteine desiderate sono assemblate in strutture amiloidi. È possibile che aggregati extracellulari amorfi possano contribuire ai risultati di legame CR, ELISA a cellule intere e MALDI. Per confermare ulteriormente la presenza di nanofibre curli ed escludere aggregati extracellulari non strutturati nei biofilm BIND, abbiamo ampiamente analizzato l'ultrastruttura dei biofilm curli utilizzando SEM ( Fig. 2f–s ) e TEM ( Supplementare Fig. 1). Per i trasformanti CR-positivi, sono state osservate sottili nanofibre associate alle cellule, che sono simili nella morfologia alla CsgA wild-type. L'analisi TEM ad alto ingrandimento delle nanofibre BIND ha rivelato che hanno un diametro di 4-7 nm, coerente con quello precedentemente riportato per le nanofibre curli 36 ( Figura 2 supplementare ). Le nanofibre BIND hanno mostrato una caratteristica morfologia aggrovigliata e sono state osservate essere strettamente associate alla superficie cellulare o talvolta esistenti come masse fluttuanti. Non sono state osservate fibre extracellulari né per il controllo plasmidico vuoto né per il biofilm Mms6-BIND ( Fig. 2f,s e Fig. 1a,n supplementare), confermando la mancanza di colorazione CR e segnali ELISA a cellule intere per questi campioni. Abbiamo inoltre eseguito l'etichettatura immunogold dei biofilm BIND che producono la proteina di fusione CsgA-FLAG, utilizzando un anticorpo anti-FLAG. Le immagini TEM immunogold mostrano la localizzazione delle nanoparticelle d'oro nei grovigli di fibre curli, confermando sia la presenza che l'accessibilità del dominio peptidico FLAG ( Figura 5 supplementare). In sintesi, gli esperimenti di colorazione CR e di birifrangenza CR dimostrano la presenza di amiloide extracellulare, i dati ELISA di filtrazione delle cellule intere indicano che le fusioni CsgA sono presenti come assemblaggi extracellulari, l'analisi MALDI conferma la presenza di materiale extracellulare insolubile SDS correlato in massa alle proteine di fusione previste e l'imaging al microscopio elettronico dei biofilm BIND fornisce la verifica ultrastrutturale della morfologia nanofibrillare del BIND ECM. Pertanto, i peptidi di sequenza e funzione arbitrarie possono essere visualizzati in modo efficiente come fusioni C-terminali a CsgA per l'autoassemblaggio extracellulare in nanofibre curli funzionalizzate.
Il vero valore del sistema BIND sta nella sua capacità di funzionare come un ampio biomateriale interfacciale la cui funzione può essere programmata geneticamente in modo modulare. Come dimostrazione di alcune di queste capacità, abbiamo selezionato tre peptidi dalla Tabella 1(FLAG, A3, MBD e SpyTag) con diverse funzioni e testato la loro capacità di introdurre queste nuove funzioni nei biofilm che producono curli. In particolare, abbiamo studiato la capacità di programmare la biotemplazione di nanoparticelle inorganiche (A3), l'adesione potenziata alle superfici abiotiche (MBD) e l'immobilizzazione covalente di proteine a lunghezza intera nei biofilm BIND (SpyTag). Abbiamo anche esaminato la generazione di biofilm BIND multifunzionali (FLAG e SpyTag). Per questi studi funzionali, abbiamo scelto un ceppo cellulare diverso come telaio per una robusta produzione di curli, un mutante di delezione di csgA precedentemente sviluppato del ceppo PHL628 di E.coli K-12 (rif. 37). Sebbene producano anche cellulosa, il che le rende inadatte alla caratterizzazione iniziale della piattaforma BIND, le cellule PHL628 (MG1655 malA-Kan ompR234 ) sono superiori al ceppo LSR10 per la produzione di curli a causa di una mutazione puntiforme nella proteina OmpR, che migliora l'espressione dell'intero operone curli di ~3,5 × , determinando una notevole quantità di produzione di curli 38 . Questo fenotipo è ideale per generare un'ampia ECM ricca di amiloide per l'analisi funzionale; sovraespressione basata su plasmide di varianti eterologhe di BIND in un csgAÈ probabile che il mutante knockout PHL628 (di seguito denominato semplicemente PHL628) prevenga l'accumulo delle proteine di fusione a livello intracellulare fornendo un'elevata produzione basale delle proteine necessarie per l'elaborazione e la secrezione efficienti delle fusioni CsgA. L'espressione dell'intera libreria di peptidi BIND in cellule PHL628 ha portato a modelli di produzione di ricci relativi simili come determinato dal legame CR in confronto al ceppo LSR10 ( Figura 6 supplementare ). Abbiamo anche eseguito ELISA di filtrazione su cellule intere sui biofilm FLAG-, A3-, SpyTag- e MBD-BIND per dimostrare che la proteina CsgA viene esportata e si assembla in un materiale extracellulare ad alto peso molecolare ( Figura 7a,c, supplementare). per esempio). Inoltre, l'analisi MALDI-TOF/TOF del materiale extracellulare purificato insolubile in SDS mostra picchi di massa che corrispondono a quelli delle proteine di fusione attese, suggerendo che sono presenti e non proteolizzate come fibrille amiloidi nell'ECM ( Figura supplementare 7b,d, f, h ).
Nanoparticelle d'argento modellate su nanofibre A3 BIND
I peptidi ingegnerizzati funzionalizzati sull'ECM possono anche essere utilizzati per promuovere i modelli di materiali, che dimostriamo utilizzando BIND composto da una fusione CsgA-A3 prodotta nelle cellule PHL628. Il peptide A3 è stato precedentemente sviluppato da phage display per legare l'argento ed è stato dimostrato che controlla la modellazione delle nanoparticelle d'argento 25 . Il biofilm di CsgA di tipo selvatico non ha modellato in modo apprezzabile le nanoparticelle d'argento ( Fig. 3a ). Al contrario, i biofilm A3-BIND mostrano una maggiore capacità di legarsi alle nanoparticelle d'argento in crescita da una soluzione di AgNO 3 , come mostrato da immagini TEM rappresentative di A3-BIND incubate che mostrano l'assemblaggio di nanoparticelle attraverso le nanofibre ( Fig. 3b, c ). Questi risultati sono riproducibili ( Fig. 8 supplementare) e dimostrare l'utilità di matrici di biofilm programmabili per la modellazione e l'organizzazione di nanoparticelle per formare nanofili unidimensionali. Le nanofibre decorate con nanoparticelle risultanti mostrano una sorprendente somiglianza con le fibre extracellulari naturali che riducono i metalli di Geobacter sulfurreducens , che hanno dimostrato di essere elettricamente conduttive 39 , suggerendo che la biotemplazione basata su BIND può essere una strategia praticabile per il de novo su larga scala produzione di nanofili conduttivi.
Le nanoparticelle d'argento sono state modellate da biofilm A3-BIND incubati in AgNO 3 acquoso . Micrografie TEM rappresentative dimostrano che le cellule PHL628 Δ csgA che producono CsgA wild-type ( a ) non mostrano modelli di nanoparticelle , mentre A3-BIND ( b ) modella le nanoparticelle dopo l'incubazione in 147 mM AgNO 3 per 4 h. ( c ) Viene mostrato un maggiore ingrandimento delle nanoparticelle di Ag organizzate su nanofilamenti A3-BIND. Tutte le barre della scala sono 0,5 μm.
(continua su link).
BANCA DATI MORGELLONS Video -CURE-Brevetti - Documenti |