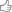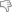-
|
| .
|
|
|
|
DIPA-CRISPR : Editing Genetico degli insetti
https://pubmed.ncbi.nlm.nih.gov/35637909/
DIPA-CRISPR è un metodo semplice e accessibile per l'editing genetico degli insetti

Astratto
Gli attuali approcci per l'editing genetico degli insetti richiedono la microiniezione di materiali negli embrioni precoci. Ciò limita fortemente l'applicazione dell'editing genetico a un gran numero di specie di insetti, in particolare a quelle i cui sistemi di riproduzione precludono l'accesso agli embrioni precoci per l'iniezione. Per superare queste limitazioni, riportiamo un metodo semplice e accessibile per l'editing genetico degli insetti, denominato CRISPR "parentale diretto" (DIPA-CRISPR). Mostriamo che l'iniezione di ribonucleoproteine Cas9 (RNP) nell'emocoel di femmine adulte introduce in modo efficiente mutazioni ereditabili nello sviluppo di ovociti. È importante sottolineare che la proteina Cas9 standard disponibile in commercio può essere utilizzata direttamente per DIPA-CRISPR, il che rende questo approccio altamente pratico e fattibile. DIPA-CRISPR consente un editing genetico altamente efficiente negli scarafaggi, sui quali non è possibile applicare approcci convenzionali, e nel coleottero modello Tribolium castaneum . Grazie alla sua semplicità e accessibilità, DIPA-CRISPR estenderà notevolmente l'applicazione della tecnologia di editing genetico a un'ampia varietà di insetti.
Parole chiave: CRISPR-Cas9; DIPA-CRISPR; coleotteri; scarafaggi; editing genetico; modifica del genoma.

Figure 1 Editing genetico di scarafaggio con DIPA-CRISPR (A) Ninfe appena nate (punte di freccia) dall'ooteca (freccia) di Blattella germanica . (B) Iniezione per adulti in B. germanica . (C) Gli occhi del mosaico di cinabro G 0 ninfe con perdita dei pigmenti dell'occhio nero. Barre di scala, 100 μm. (D) Un risultato rappresentativo della genotipizzazione del cinabro G 0 . Le ninfe G 0 con occhi normali nate da una singola ooteca sono state analizzate mediante test di mobilità eteroduplex (HMA). Sono indicati gli individui con alleli modificati (asterischi) e le bande omoduplex (156 bp, punta di freccia). Vedere anche la Figura S1 per i risultati dettagliati. (E) Efficienza di editing genetico (GEF) di DIPA-CRISPR. I mutanti G 0 sono stati prima sottoposti a screening per fenotipi (bianchi oa mosaico), quindi quelli senza fenotipi sono stati analizzati mediante HMA (genotipizzazione). Bianco, con fenotipi in intere regioni di entrambi gli occhi; AO, dopo la caduta dell'ooteca; EER, presenza o assenza di clorochina (2 mM) nella soluzione iniettabile.

Ereditarietà degli alleli modificati negli scarafaggi (A e B) Uno schema di accoppiamento (A) ei risultati della genotipizzazione (B) degli G 1 individui . I due mosaici di cinabro mostrati nella Figura 1C (G 0 #1 e #2) sono stati incrociati con il tipo selvaggio e tutta la G 1 progenie è stata analizzata individualmente mediante test di mobilità eteroduplex. Gli alleli mutanti sono indicati sotto i pannelli (a–g, sette alleli in totale). Vedere la Figura S1D per le loro sequenze nucleotidiche. (C) Uno schema di accoppiamento per lo screening degli insetti knockout G 1 . Insetti G0 progenie con fenotipi del colore degli occhi sono stati incrociati per ottenere . G1 (D) Un di cinabro adulto knockout G 1 (a destra). Le punte di freccia indicano gli occhi bianchi. Barre di scala, 1 mm. (E) Fenotipi negli insetti G 1 . Ogni riga indica il risultato di un accoppiamento a coppia singola di G 0 .
|
|
|
| .
|
0 replies since 11/8/2023, 15:56 13 views
.